清华大学生命学院孙前文实验室在《植物细胞》发文
2020-02-22 03:30:162020年2月19日,清华大学生命科学学院孙前文实验室在《植物细胞》(The Plant Cell)发表题为“拟南芥不同发育时期和胁迫响应下的R-loop图谱”(The R-loop atlas of Arabidopsis development and responses to environmental stimuli)的研究论文。
R-loop是一种由单链DNA和DNA:RNA杂合链组成的三链核酸结构。已有的研究表明,R-loop广泛存在于各个物种中,并且在染色质修饰、基因表达调控、DNA损伤修复、染色体分离和基因组稳定性等生物学过程中发挥重要的功能。2017年,孙前文实验室开发了高效的全基因组R-loop检测技术--ssDRIP-seq(ssDRIP-seq, single-strand DNA ligation-based library construction of DNA:RNA hybrid immunoprecipitation and sequencing图1,详见报道3,原文链接5,),并首次报道了模式植物拟南芥幼苗期的R-loop图谱。在此工作基础上,孙前文实验室进一步绘制了拟南芥在不同发育时期以及各种胁迫响应下的R-loop图谱,并揭示了拟南芥R-loop特有的动态变化特征。
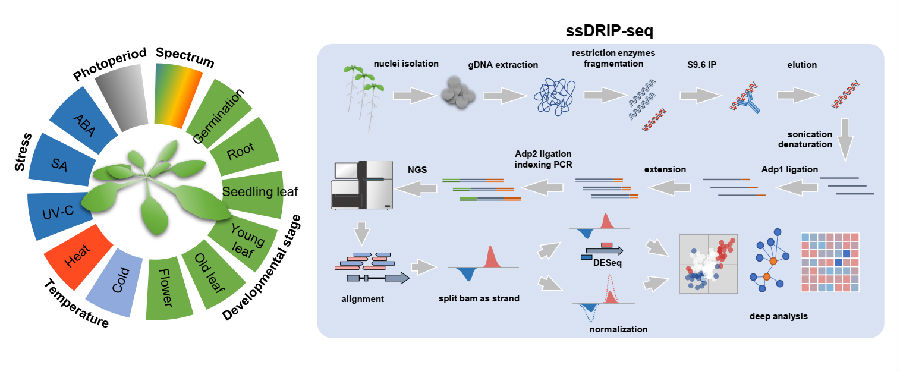
图1 ssDRIP-seq技术流程以及本研究涉及的拟南芥材料
该工作共研究了53个拟南芥材料(图1),主要包括:不同生理发育时期(萌发苗、根、苗叶、花、新叶和老叶);不同的光照,光周期和温度处理(蓝光、红光、远红光;黑暗、长日照、短日照、全日照;4℃、17℃、22℃、27℃、37℃);不同的激素和逆境处理(脱落酸ABA、水杨酸SA以及UV-C紫外光照射)。分析后发现,在不同的营养生长时期、光照或胁迫响应下,R-loop在基因组上的分布并无显着差异,这些发育时期及胁迫处理的R-loop模式基本一致(图2A)。但在花和萌发幼苗,以及长时间37℃热处理样品中,R-loop变化差异较大(图2A)。进一步分析发现基因组中各个基因的转录区正义R-loop(sGB-R-loop)和转录起始位点反义R-loop(asTSS-R-loop)在不同样本中的变化并没有表现出显着的偶联(图2B),表明这两类R-loop有着不同的调控机制。此外,他们发现基因上的R-loop的水平和对应的RNA的表达量也没有呈现显着的偶联(图2C),证明R-loop的动态变化有着相对独立的调控机制,并不能被简单地看作是RNA表达水平动态变化的副产物。这一研究首先在拟南芥中对R-loop在不同的生理发育时期和胁迫响应下进行表征分析,大量数据积累和分析能够有力的支持和促进后续的植物R-loop生物学功能的研究。该工作共研究了53个拟南芥材料(图1),主要包括:不同生理发育时期(萌发苗、根、苗叶、花、新叶和老叶);不同的光照,光周期和温度处理(蓝光、红光、远红光;黑暗、长日照、短日照、全日照;4℃、17℃、22℃、27℃、37℃);不同的激素和逆境处理(脱落酸ABA、水杨酸SA以及UV-C紫外光照射)。分析后发现,在不同的营养生长时期、光照或胁迫响应下,R-loop在基因组上的分布并无显着差异,这些发育时期及胁迫处理的R-loop模式基本一致(图2A)。但在花和萌发幼苗,以及长时间37℃热处理样品中,R-loop变化差异较大(图2A)。进一步分析发现基因组中各个基因的转录区正义R-loop(sGB-R-loop)和转录起始位点反义R-loop(asTSS-R-loop)在不同样本中的变化并没有表现出显着的偶联(图2B),表明这两类R-loop有着不同的调控机制。此外,他们发现基因上的R-loop的水平和对应的RNA的表达量也没有呈现显着的偶联(图2C),证明R-loop的动态变化有着相对独立的调控机制,并不能被简单地看作是RNA表达水平动态变化的副产物。这一研究首先在拟南芥中对R-loop在不同的生理发育时期和胁迫响应下进行表征分析,大量数据积累和分析能够有力的支持和促进后续的植物R-loop生物学功能的研究。
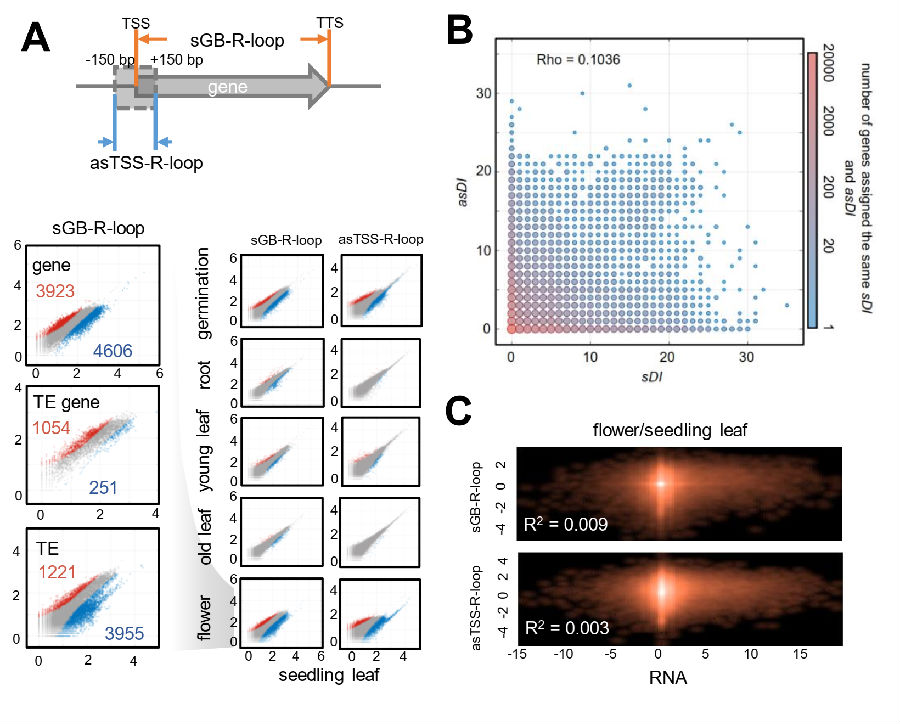
图2 R-loop动态变化的主要结果
清华大学生命学院孙前文课题组博士后徐炜、博士生李宽、李帅、以及博士后侯全璨为本文共同第一作者,博士后张禹舜和博士后刘坤朋参与部分数据分析和相关实验,孙前文研究员为本文的通讯作者。该工作得到国家自然科学基金委、科技部国家重点研发计划以及生命科学联合中心等经费的支持。
论文链接:http://www.plantcell.org/content/early/2020/02/19/tpc.19.00802
相关推送报道:
1. 孙前文实验室在Cell Reports发文报道R-loop介导拟南芥中叶绿体基因组稳定性维持的新机制
http://www.cls.edu.cn/Research/Research_Achievements4851.shtml
2. 孙前文实验室The Plant Cell发文报道R-loop影响叶绿体基因组稳定的调控机制
http://www.cls.edu.cn/Research/Research_Achievements3897.shtml
3. 孙前文研究组与杨雪瑞研究组合作发文报道精准、高效、高通量检测R-loop分布的新方法
http://www.cls.edu.cn/Research/Research_Achievements3861.shtml
4. 清华孙前文组等揭示水稻根发育及向地性的新机制
http://www.sohu.com/a/134412727_650136
相关原文链接:
1.Xu W, Li K, Li S, Hou Q, Zhang Y, Liu K, Sun Q. The R-loop Atlas of Arabidopsis Development and Responses to Environmental Stimuli. Plant Cell. 2020 Feb; tpc.00802.2019.
http://www.plantcell.org/content/early/2020/02/19/tpc.19.00802
2. Yang Z, Li M, Sun Q. RHON1 Co-transcriptionally Resolves R-Loops for Arabidopsis Chloroplast Genome Maintenance. Cell Rep. 2020 Jan 7;30(1):243-256.e5. doi: 10.1016/j.celrep.2019.12.007.
https://www.cell.com/cell-reports/fulltext/S2211-1247(19)31646-8
3. Yuan W, Zhou J, Tong J, Zhuo W, Wang L, Li Y, Sun Q, Qian W. ALBA protein complex reads genic R-loops to maintain genome stability in Arabidopsis. Sci Adv. 2019 May 15;5(5):eaav9040. doi: 10.1126/sciadv.aav9040.
https://advances.sciencemag.org/content/5/5/eaav9040.full
4. Yang Z, Hou Q, Cheng L, Xu W, Hong Y, Li S, Sun Q. RNase H1 Cooperates with DNA Gyrases to Restrict R-Loops and Maintain Genome Integrity in Arabidopsis Chloroplasts. Plant Cell. 2017 Oct;29(10):2478-2497. doi: 10.1105/tpc.17.00305.
http://www.plantcell.org/content/early/2017/09/21/tpc.17.00305?rss=1
5. Xu W, Xu H, Li K, Fan Y, Liu Y, Yang X, Sun Q. The R-loop is a common chromatin feature of the Arabidopsis genome. Nat Plants. 2017 Sep;3(9):704-714. doi: 10.1038/s41477-017-0004-x.
https://www.nature.com/articles/s41477-017-0004-x
6. Shafiq S, Chen C, Yang J, Cheng L, Ma F, Widemann E, Sun Q. DNA Topoisomerase 1 Prevents R-loop Accumulation to Modulate Auxin-Regulated Root Development in Rice. Mol Plant. 2017 Jun 5;10(6):821-833. doi: 10.1016/j.molp.2017.04.001.
http://www.cell.com/molecular-plant/fulltext/S1674-2052(17)30102-8
7. Sun Q, Csorba T, Skourti-Stathaki K, Proudfoot NJ, Dean C. R-loop stabilization represses antisense transcription at the Arabidopsis FLC locus. Science. 2013 May 3;340(6132):619-21.
https://science.sciencemag.org/content/340/6132/619.long

